Menneske-sjimpanse likhet i genomet
(Fritt etter 'More than a Monkey', Jeffrey Tomkins, PhD; Kap. 3)
Ønsker du en kjapp (9m:30s), visuell gjennomgang, kan denne videoen være av interesse.
Utenom det allerede nevnte (kap2), så er objektive sammenlikninger av genom meget sjeldne. Det har riktignok vært noen rapporter som er verd å merke seg, når vi prøver å få et mer pålitelig tall for genom-likhet mellom sjimpanser og mennesker. Vi skal også se på en simulering, kjørt ut fra åpent tilgjengelige databaser og programvare.
Tilfeller av ulikhet mellom sjimpanse og menneske
På internett hender det at det dukker opp anonyme innlegg. Ofte er bakgrunnen tvilsom, men i tilfelle evolusjonsteorien, hvor det å melde opposisjon og tvil i forhold til teorien, kan resultere i betydelig reduserte muligheter for avansement etc, kan det etter min mening forstås. Et slikt tilfelle dukket opp i et profesjonelt journalist-format. Det som gjorde rapporten troverdig, var at den ga tilgang til scriptet (programvare kode), fritt tilgjengelig på web-stedet i tillegg til en klar referanse til deres offentlig benyttede datakilder. Genom-dataene som de benyttet kan fritt lastes ned her. Programvaren de benyttet var skrevet i programmeringsspråket Pearl, og kan leses og analyseres av bio-informatikk-spesialister, for å se hva som ble utført. Så eksperimentet kunne i motsetning til mange, repeteres og testes, basert på dataene de forsynte.
Nevnte artikkel benyttet en algoritme som involverte tilfeldig utvelgelse av 10.000 30-basers sekvenser fra hvert sjimpanse kromosom, og så bestemte likhet og samsvar med den sekvensen sammenlignet med motparten i menneskelig kromosom. Dette studiet kom opp med en gjennomsnittlig DNA-likhet på 88,5%. Mens tilnærmingen i dette studiet var nytt og objektivt, så involverte det bare tilfeldig utvalg av et begrenset mengde på 10.000 av små kromosom-biter fra hvert sjimpanse kromosom i genomet. Likevel var det et nytt og forfriskende innslag i all håndplukking av data, for å få så høy likhet som mulig.
I 2011 startet forfatteren av denne boka en rapport for å bane vei for tilleggsforskning som kunne hjelpe til å definere helhetlig genom-likhet mellom sjimpanser og mennesker (2). Prosjektet startet med å laste ned data som i prinsippet skulle være tilfeldige DNA-sekvenser (40.000) fra sjimpanse genomet, via National Center for Biotechnology (NCBI). Kommunikasjon med staben ga imidlertid informasjon at dataene var utvalgt og pakket sammen, fordi de skulle være mest mulig lik mennesket. Selv om det altså ikke var tilfeldige data, måtte forfatteren ta de data han kunne få. Han undersøkte så disse sjimpanse-data med fire ulike versjoner av det menneskelige genom, ved hjelp av BLASTN algoritmer, med varierende matchende parametre. I det studiet var graden av likhet 86-89%, avhengig av parameterverdier benyttet. De algoritme-parameterverdier som ga lengste segmenter av matcher, oppnådde bare 86% likhet med mennesket. Dessuten fant en at kromsomer måtte deles i biter (inntil 740 baser), for at ikke programmet skulle stoppe på statistiske terskelverdier.
En større undersøkelse om sjimpanse-genomet
Da Forfatteren fulgte opplegget i studiet nevnt ovenfor, brukte han de foreløpige dataene det forsynte, til å sette opp en større undersøkelse. Han gikk systematisk til verks, f.eks. for sjimpanse kromosom nr.1, delte han dataene i porsjoner' på 150 base-segmenter, ut fra en grense på 740 i første undersøkelse. Så øket han antallet med 50 pr sammenligning, og så hvilket antall som ga den høyeste match med menneskelig kromosom 1. Dette ble utført for hvert sjimpanse kromosom, siden det er forskjeller dem i mellom i forhold til mennesket.
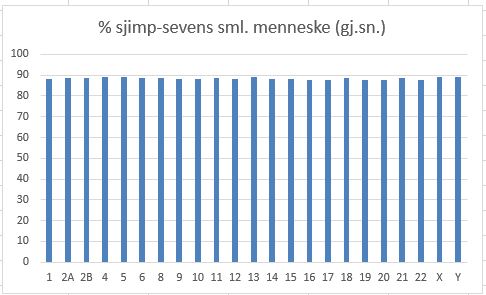 Testen tok ikke i beregning menneskelig DNA som ikke er med i sjimpanse, heller ikke en liten rest sjimpanse-DNA som ikke er henført til ett bestemt kromosom (unanchored contigs). Det ville medført en enda lavere genom likhet. Data er samlet i Bilde 2, mer spesifikke data her. Kromosom 2A og 2B hos sjimpanse ble sammenlignet med kromosom 2 hos mennesket. For genomet som helhet var det en gjennomsnittlig likhet på 88,5%.
Testen tok ikke i beregning menneskelig DNA som ikke er med i sjimpanse, heller ikke en liten rest sjimpanse-DNA som ikke er henført til ett bestemt kromosom (unanchored contigs). Det ville medført en enda lavere genom likhet. Data er samlet i Bilde 2, mer spesifikke data her. Kromosom 2A og 2B hos sjimpanse ble sammenlignet med kromosom 2 hos mennesket. For genomet som helhet var det en gjennomsnittlig likhet på 88,5%.
Bilde 1. Resultat ut fra større undersøkelse
Konklusjon: Sjimpanse og menneske DNA ikke så like som noen vil ha det til.
Sjimpanse og menneske deler mange lokaliserte proteinkodende regioner, fra høy til moderat DNA-likhet. Imidlertid er de hva som forventes av to ulike og separat skapte slags organismer.
Analysene utført ved hjelp av BLASTN v 2.2.25 + og nucmer indikerer at de justerbare delene av gjeldende sjimpanse-genomdannelsesenheten er ca. 88% lignende i gjennomsnitt til menneske. Detaljert resultat og problemstillinger knyttet til hver analyse diskuteres under, gjort rede for av forfatteren her:
Det har skjedd endringer i resultatene her i okt. 2015, grunnet dette:
En feil eller uregelmessighet i BLASTN-algoritmen eksisterer tydelig i versjoner 2.2.26+ til 2.2.30+ (fem påfølgende utgivelser) som utelater signifikante deler av ikke-lignende spørringssekvens under de betingelser som benyttes i denne studien. Gitt at den eneste tilsynelatende intakte versjonen av BLASTN-algoritmen ble testet var 2.2.25 +, ble hele 2013-studien som tidligere ble publisert av denne forfatter gjentatt med denne versjonen ved hjelp av et fragmentskive på 300 baser for hvert kromosom som produserte et gjennomsnittlig overordnet genom likhet på ca. 88% identitet. Interessant var dette resultatet innenfor rekkevidden av justeringsidentiteter (86 til 89%) oppnådd i en foreløpig studie utført av denne forfatteren ved bruk av 40.000 tilfeldige chimpanse-spor-leser som ble forespurt mot seks forskjellige versjoner av det menneskelige genomet også ved bruk av versjon BLASTN versjon 2.2. 25+ (Tomkins 2011a). Det skal imidlertid bemerkes at disse ikke er identiske sett med kode, siden programvareoppdateringer har blitt utført til BLASTN v2.2.25 sammenlignet med versjonen som ble brukt av denne forfatteren i 2011.
Sammendrag
Oppsummert kan det være greit å si at sjimpanse-genometet ikke er 98 til 99% lik menneskelig, men ikke mer enn 88,5% likt generelt. Det er imidlertid flere forbehold som må vurderes. For det første ble sjimpansens genomiske sekvens som ble brukt i denne studien samlet med det menneskelige genom som et rammeverk og står således ikke for egne verdier (Tomkins 2011b). For det andre indikerer flertallet av strømnings-cytometriundersøkelser av sjimpanse DNA-kjerner sammen med den cytogenetiske analysen av kromosomer en genomsnittlig størrelsesforskjell på ca. 8%, med sjimpansegenomet som har en signifikant større mengde heterochromatisk DNA sammenlignet med mennesker (Formenti et al., 1983 Pellicciari et al., 1982, 1988, 1990a, 1990b; Seuanez et al., 1977).
Myten om fusjon av kromosom 2
(Fritt etter 'More than a Monkey', Jeffrey Tomkins, PhD; Kap. 4)
Ett av de førende argumenter for at mennesker skal ha utviklet seg fra aper, er kjent som kromsom 2 fusjons-argumentet. Denne hypotetiske idéen går ut på en ende-til-ende fusjon av to små apelike kromosomer (2A og 2B) produserte menneskets kromosom 2. Dette postulerte scenarioet skal visstnok forklare forskjellen i antall kromosomer hos mennesket 46=23*2 og hos aper (sjimpanser, gorillaer og orangutanger): 48=24*2.
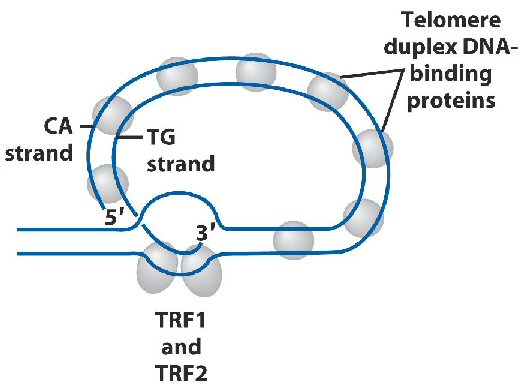 Opprinnelig idé for fusjonsmodellen var basert på delt bindings-mønstre observert under lys-mikroskopi for kjemisk tilsmussede kromosomer. Prosessen er basert på isolering av celler som gjennomgår kondensering under mitose (celledeling), som tilsmusses med et stoff som binder til DNA, og produserer bindemønstre (karyotyping). Da dette ble gjort for sjimpanser, var det mange små kromosomer som delte mange band lik menneskets kromosom 2, og ble deretter postulert å ha fusjonert til å forme kromosom 2 hos mennesket. Det var en størrelsesforskjell på ca. 10% eller 24 millioner baser i den foreslåtte fusjonen. Vi kjenner nå til at de tilsmussede bandene en refererte til biokjemisk, tilsvarer GC og AT %-andeler i DNA, ikke spesifikke gener eller gen-grupper. De mange genene i området som omgir den antatte fusjonen, utgjør 614.000 baser som ikke finnes i sjimpanse, og deler ikke komparativ likhet med kromsom 2A eller 2B hos sjimpanse.
Opprinnelig idé for fusjonsmodellen var basert på delt bindings-mønstre observert under lys-mikroskopi for kjemisk tilsmussede kromosomer. Prosessen er basert på isolering av celler som gjennomgår kondensering under mitose (celledeling), som tilsmusses med et stoff som binder til DNA, og produserer bindemønstre (karyotyping). Da dette ble gjort for sjimpanser, var det mange små kromosomer som delte mange band lik menneskets kromosom 2, og ble deretter postulert å ha fusjonert til å forme kromosom 2 hos mennesket. Det var en størrelsesforskjell på ca. 10% eller 24 millioner baser i den foreslåtte fusjonen. Vi kjenner nå til at de tilsmussede bandene en refererte til biokjemisk, tilsvarer GC og AT %-andeler i DNA, ikke spesifikke gener eller gen-grupper. De mange genene i området som omgir den antatte fusjonen, utgjør 614.000 baser som ikke finnes i sjimpanse, og deler ikke komparativ likhet med kromsom 2A eller 2B hos sjimpanse.
Bilde 2. Telomerer som protein-bindinger -se også her.
Det antatte fusjonsstedet
 Det påståtte stedet for den evt. fusjonen er foreslått til kromosom 2, og DNA-segmentet ble først lokalisert og sekvensiert i 1991, omkring 9 år etter fusjonen først ble foreslått ved sammenlignende studier. Men den antatte fusjonen ble noe av en overraskelse for forskere, av det som syntes som en korrupt telomer fusjon. Ut fra hva de kjente av fusjonsdata fra levende pattedyr, hadde de forventet noe annet enn dette. Også tapet av 10% informasjon, var vanskelig å forklare.
Det påståtte stedet for den evt. fusjonen er foreslått til kromosom 2, og DNA-segmentet ble først lokalisert og sekvensiert i 1991, omkring 9 år etter fusjonen først ble foreslått ved sammenlignende studier. Men den antatte fusjonen ble noe av en overraskelse for forskere, av det som syntes som en korrupt telomer fusjon. Ut fra hva de kjente av fusjonsdata fra levende pattedyr, hadde de forventet noe annet enn dette. Også tapet av 10% informasjon, var vanskelig å forklare.
Bilde 3. Hypotetisk kromosom 2-fusjon
Bilde 3-kommentar: Avbildning av hypotetisk scenario der sjimpanse kromosom 2A og 2B fusjonerer ende til ende, og danner menneskelig kromosom 2.Merk den ekstreme mangel på posisjonerings samsvar for det kryptiske sentromer-stedet og størrelsesulikheten som utgjør ca. 10%, eller 24 millioner baser.
Om det antatte fusjonsstedet faktisk indikerte en ende-til-ende kromosomfusjon, ville det være den første slike der pattedyr var involvert. Det skyldes at alle dokumenterte kjente pattedyr kromosom-fusjoner i levende dyr involverer en spesifikk sekvens, kalt satelitt_DNA (satDNA). Denne er kjent over hele genomet, og spesielt rundt sentromerer, der de fleste krosomer deles før de evt. fusjonerer. Nærværet av satDNA er et nøkkelkjennetegn som innbefatter det initielle bruddet og påfølgende fusjonerte sekvens, som er observert i alle studerte fusjoner funnet i pattedyr. Kromosom-fusjoner som inneholder hypotetiske telomer-telomer DNA-signaturer er ikke tidligere dokumentert. Det skyldes bl.a. at telomerer inneholder høyt spesialiserte beskyttere som hindrer fusjon, som heller ikke er observert i naturen. Også 'vanlige' kromosom-fusjoner som innbefatter satDNA er ekstremt sjeldne begivenheter.
Ved enden på alle lineære kromosomer er spesielle DNA sekvenser, kalt telomerer. I mennesker og pattedyr, består telomerer normalt av tusener av repetisjoner av en 6-baset sekvens: "TTAGGG". Typisk menneskelige telomerer inneholder 1.700-2.500 av diss seks-basers repetisjoner i perfekt to-spann (6). Så spørsmålet blir: 'Har 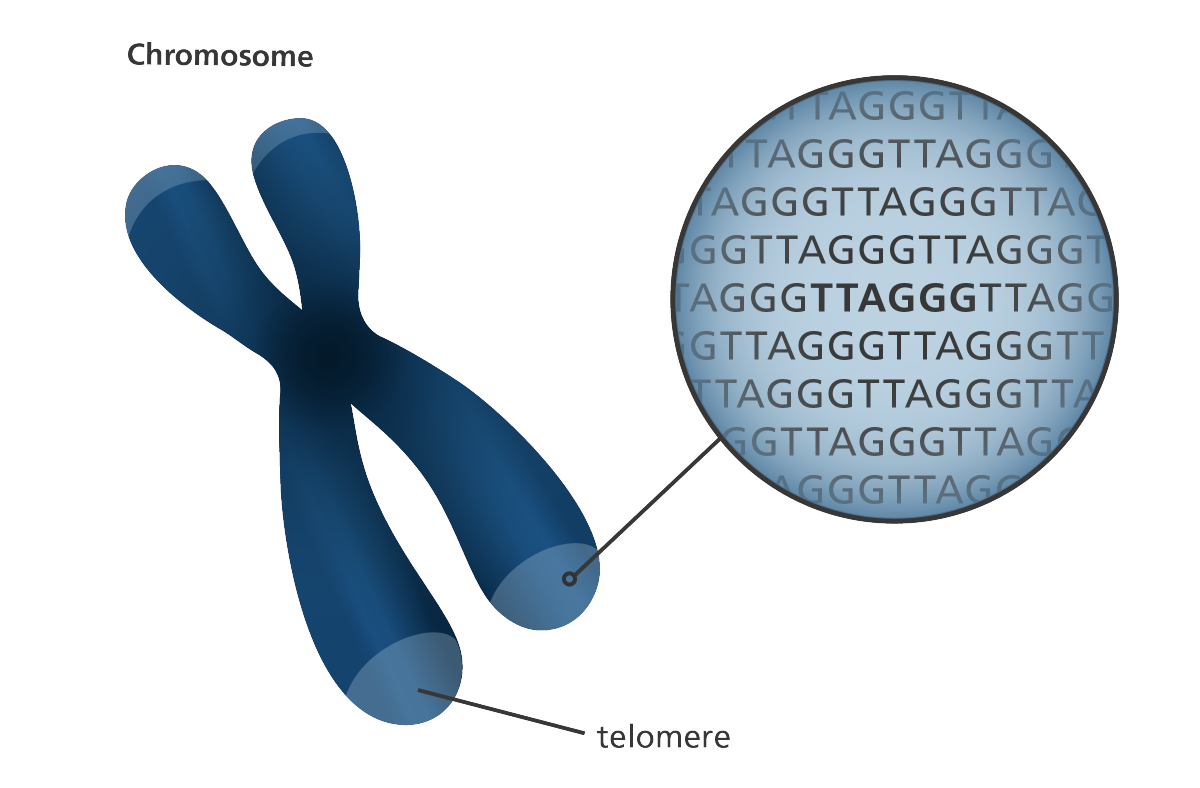 den hypotetiske kromosom 2-fusjonen de forventede karakteristika av en ende-til-ende fusjon for to kromosomer? For å si det kort: dataene passer ikke. Sekulære evolusjonsforskere har uttalt dette faktum i sine forsknings rapporter (se under). Kanskje en av de mest avslørende var en nøye DNA-sekvens analyse, av fusjonsområdet av molekyl-genetikere, der de merket seg at den fusjonerte DNA-sekvensen var ekstremt degenerert, ut fra sitt hypotetiske opphav. 6 millioner år er overgang fra primater til menneske antatt å vare. I det store tidsdypet, er det kun en kort periode. Sammenlignet med en opprinnelig fusjon av samme signatur-størrelse 8798 baser), så er det formodede fusjonsstedet bare ca. 88% likt. Noen har kommentert dette slik: "hode-til-hode arrayer av repetisjoner ved fusjonsstedet har degenert betydelig, fra de nær perfekte arrayene (TTAGGG) som finnes i telomerer" Og: "om fusjonen inntraff innen telomer-repeterende arrayer, for mindre enn 6 millioner år siden: hvorfor er arrayene på fusjonsstedet så degenererte?
den hypotetiske kromosom 2-fusjonen de forventede karakteristika av en ende-til-ende fusjon for to kromosomer? For å si det kort: dataene passer ikke. Sekulære evolusjonsforskere har uttalt dette faktum i sine forsknings rapporter (se under). Kanskje en av de mest avslørende var en nøye DNA-sekvens analyse, av fusjonsområdet av molekyl-genetikere, der de merket seg at den fusjonerte DNA-sekvensen var ekstremt degenerert, ut fra sitt hypotetiske opphav. 6 millioner år er overgang fra primater til menneske antatt å vare. I det store tidsdypet, er det kun en kort periode. Sammenlignet med en opprinnelig fusjon av samme signatur-størrelse 8798 baser), så er det formodede fusjonsstedet bare ca. 88% likt. Noen har kommentert dette slik: "hode-til-hode arrayer av repetisjoner ved fusjonsstedet har degenert betydelig, fra de nær perfekte arrayene (TTAGGG) som finnes i telomerer" Og: "om fusjonen inntraff innen telomer-repeterende arrayer, for mindre enn 6 millioner år siden: hvorfor er arrayene på fusjonsstedet så degenererte?
Bilde 4. Telomerer som stopp-signaler
I atskillige rapporter bemerker forskere at det hypotetiske fusjonsstedet er lokalisert i et området av menneskelig genom med små nivåer av rekombinasjoner, i og med at det er nær en sentromer. Det innebærer at om en fusjon inntraff, så ville den bære preg av svært lite degenerasjon, nesten opprinnelig burde den være (7-9). Om vi studerer fusjonsområdet nærmere, finner vi at det mest sannsynlige fusjonsområdet er 798 baser i størrelse. Det er et uventet lite DNA-segment, i forhold til om en fusjon fant sted der i fortiden. Selv om to meget små telomerer fusjonerte, ville en fusjonsregion på ca. 10.000 baser være til stede. Om de var større, ville fusjonsområdet være ca. 30.000 baser. 798 basers fusjonsområde er en meget  liten signatur, for telomer-sekvensen.
liten signatur, for telomer-sekvensen.
Et iøynefallende trekk ved fusjonsstedet er at det er en blendende mangel på intakte repeterende telomerer, og de som kan finnes eksisterer mest som uavhengige enheter, ikke som vanlig i tospann (tandem). Basert på den forutsatte modellen, skulle tusener av intakte motiv i perfekt tospann eksistere. Innen fusjonssekvensen på 798 baser, er det bare 10 intakte 'TTAGGG' telomersekvenser og bare 42 intakte 'CCCTAA' reverse komplementer som finnes. Utenfor denne regionen (på 798 baser), så forsvinner den tette gruppering av telomersekvenser fullstendig. Men det største problemet for fusjonsområdet gjenstår:
Bilde 5. DDX11L2-genet med statistikk
Fusjonsområdet er midt i et fungerende gen!
En av de mest bemerkelsesverdige oppdagelser om den formentlige fusjonen, ble ikke bredt popularisert hovedsakelig fordi det diskrediterte hele fusjonsmodellen. Det var fordi fusjonsstedet ble funnet å være innenfor en CHLR1-lik pseudogen (DDX11L2), slik en forskningsrapport fra 2002 la fram. Men dens merkelige posisjon ble ikke skikkelig erkjent i teksten i rapporten (10). Det at det midlertidig ble betraktet som et pseudogen, var en slags dekke for det oppsiktsvekkende funnet. Men 11 år senere ble dette mer grundig undersøkt av forfatteren denne boka. Det ble for noen et bombenedslag som la hele fusjonsmodellen til hvile (11).
Siden den opprinnelige rapporten i 2002, har fusjonsregionen blitt oppdatert med forbedret DNA-sekvens beskrivelser, så vel som betydelig mengder av funksjonelt genom-data, som kan sees i UCSC genom-browseren. Fusjonsstedet er definitivt lokalisert inni det første ikke-kodende segmentet (intronet) til et funksjonelt RNA-helicase gen (det nevnte DDX11L2). Dette genet er sammensatt av minst 3 ulike variable eksoner, som produserer regulerende RNA-avskrifter av ulik lengde. Noen inneholder to og andre tre eksoner (protein-kodende segment). Dette genet er også tydelig uttrykt i minst 255 ulike celle/vevs-typer. Det er også tydelig sam-uttrykt med en variasjon av viktige topp-nivå gener, knyttet til prosesser involvert i cellesignaler i utvikling av blodceller og andre ekstracellulære omgivelser.
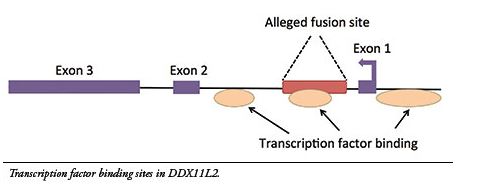 Lokaliseringen til fusjonsområdet inni et funksjonelt og høyt uttrykt gen, assosiert med uttrykk i vidt omfattende cellekategorier, benekter sterkt idéen at det er et biprodukt av en fusjon som aldri inntreffer i naturen. Komplekse, funksjonelle gener kan ikke dannes av katastrofale kromosom-fusjoner.
Lokaliseringen til fusjonsområdet inni et funksjonelt og høyt uttrykt gen, assosiert med uttrykk i vidt omfattende cellekategorier, benekter sterkt idéen at det er et biprodukt av en fusjon som aldri inntreffer i naturen. Komplekse, funksjonelle gener kan ikke dannes av katastrofale kromosom-fusjoner.
Bilde 6. Selve DDX11L2 genet
Men det stopper ikke der. Det påståtte fusjonsområdet omsetter til kode også en region innenfor DDX11L2, der avskrifts-kopier knyttes sammen -se Bilde 7. Sett fra høyre er den første ved promotoren rett ved 1.ekson og den andre er i det første intronet, samsvarende med fusjonssted-sekvensen ('inni fusjonsstedet'). Det er også en påfølgende avskrifts-kopi som følger fusjonen i det første intronet. Et slikt scenario indikerer at det påståtte fusjonsområdet er involvert i koding og regulering av DDX11L2-genet. I den reversible orienteringen, på motsatt helix-hjelper det formodede fusjonsområdet å produsere en variasjon av meget komplekse avskrifter av ulik lengde, og inneholder et nøkkel-bindingsområdet for kopierende (avskrifts)faktorer. DDX11L2 blir også regulert av mikro-RNAs. 'Fusjons-området' leses altså av cellulært nano-maskineri -i dets omvendte (reverse) rekkefølge, Så 'fusjons-området' er altså unikt posisjonert for å kontrollere den komplekse avskriften av DDX11L2-genet.
Telomerer er over hele genomet
Gjentatte telomerer, -normalt tusener av repetisjoner av en 6-baset sekvens: "TTAGGG", forekommer i store mengder over hele genomet. Tidligere trodde en at disse stedene var knyttet til kromsom-brudd, men det er nå imøtegått av nyere forskning (13). Nå mener en at disse telomerene også kan være involvert i uttrykk av gener. Det er én av de fantastisk nye oppdagelsene, at DNA-sekvenser kan ha multiple roller og funksjoner som de utøver i reguleringen av gener. Selv om det prosentvis bare er 0,25%, så inneholder bare nevnte kromosom 2 ca. 91.000 baser/nukleotider (0,23%). Det synes nå som de kan tjene flere viktige funksjoner, ved siden av deres spesifikke rolle ved endene av kromosomet.
Den såkalte kryptiske sentromeren
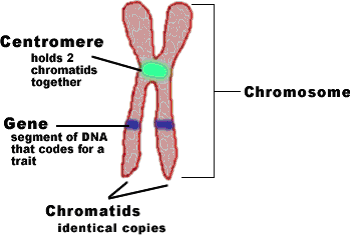 Enda et annet hovedproblem ved fusjonsmodellen er mangel på bevis for en kryptisk sentromer lokasjon. Sentromerer er den sentrale delen av et kromosom, som spiller en nøkkelrolle i kontroll ved celledeling og gen-stabilitet. Om det var en hode-til-hode fusjon av to kromosomer -som nevnt ovenfor, ville det være to centrosomer i det fusjonerte kromosomet. Nå er det dårlig nytt for et kromosom, den kan bare ha én. Én av centrosomene måtte umiddelbart elimineres, eller i det minste få stilnet funksjonen for celledeling, ellers ville kromosomet selv-destrueres. Interessant nok er tegnene på en kryptisk sentromer i kromosom 2 enda svakere enn for et telomer-rikt fusjonssted. Det kan også bemerkes at angivelig sted for en kryptisk sentromer, er på feilaktig sted, i forhold til fusjonsmodellen.
Enda et annet hovedproblem ved fusjonsmodellen er mangel på bevis for en kryptisk sentromer lokasjon. Sentromerer er den sentrale delen av et kromosom, som spiller en nøkkelrolle i kontroll ved celledeling og gen-stabilitet. Om det var en hode-til-hode fusjon av to kromosomer -som nevnt ovenfor, ville det være to centrosomer i det fusjonerte kromosomet. Nå er det dårlig nytt for et kromosom, den kan bare ha én. Én av centrosomene måtte umiddelbart elimineres, eller i det minste få stilnet funksjonen for celledeling, ellers ville kromosomet selv-destrueres. Interessant nok er tegnene på en kryptisk sentromer i kromosom 2 enda svakere enn for et telomer-rikt fusjonssted. Det kan også bemerkes at angivelig sted for en kryptisk sentromer, er på feilaktig sted, i forhold til fusjonsmodellen.
Bilde 7. Sentromer som binding for kromatider
Sentromer-sekvenser er dannet av varianter av DNA-sekvenser, som er ca. 171 baser lange. Noen typer finnes over hele genomet, mens andre er spesifikke for centomerer. Den type som finnes i den kryptiske sentromeren passer ikke i forhold til funksjonelle menneskelige sentromerer (9). Enda verre for evolusjonister, er at de heller ikke har noen motpart overhodet i sjimpanse-genomet. De aktuelle sekvensene er av en høyere ordens gjentatte telomerer, som finnes i menneskelig genom utenom sentromerer.
 Konklusjon: dårlige data gir ingen fusjon
Konklusjon: dårlige data gir ingen fusjon
Det foreslåtte DNA-beviset for en fusjon av to sjimpanse kromosomer, eksisterer ikke under nøyaktig inspeksjon av dataene. DNA-sekvensen er alt for tynn til å slutte en fusjon av telomerer. Repeterende telomerer, som finnes over hele genomet, kan spille en viktig rolle i genreguleringen. Det såkalte fusjonsstedet er lokalisert inni et fungerende og høyst komplekst RNA-gen, og det hjelper med avskrift/kopiering og å regulere gener. Det er også tynt bevis for en kryptisk sentromer, og enda mindre for at den kom fra en sjimpanse. Den overveldende bevisbyrden gir som nødvendig konklusjon at fusjonen aldri fant sted.
Bilde 8. Rett til egne meninger, ikke egne fakta
Utvalg av stoff og bilder ved Asbjørn E. Lund
 Testen tok ikke i beregning menneskelig DNA som ikke er med i sjimpanse, heller ikke en liten rest sjimpanse-DNA som ikke er henført til ett bestemt kromosom (unanchored contigs). Det ville medført en enda lavere genom likhet. Data er samlet i Bilde 2, mer spesifikke data her. Kromosom 2A og 2B hos sjimpanse ble sammenlignet med kromosom 2 hos mennesket. For genomet som helhet var det en gjennomsnittlig likhet på 88,5%.
Testen tok ikke i beregning menneskelig DNA som ikke er med i sjimpanse, heller ikke en liten rest sjimpanse-DNA som ikke er henført til ett bestemt kromosom (unanchored contigs). Det ville medført en enda lavere genom likhet. Data er samlet i Bilde 2, mer spesifikke data her. Kromosom 2A og 2B hos sjimpanse ble sammenlignet med kromosom 2 hos mennesket. For genomet som helhet var det en gjennomsnittlig likhet på 88,5%. Opprinnelig idé for fusjonsmodellen var basert på delt bindings-mønstre observert under lys-mikroskopi for kjemisk tilsmussede kromosomer. Prosessen er basert på isolering av celler som gjennomgår kondensering under mitose (celledeling), som tilsmusses med et stoff som binder til DNA, og produserer bindemønstre (karyotyping). Da dette ble gjort for sjimpanser, var det mange små kromosomer som delte mange band lik menneskets kromosom 2, og ble deretter postulert å ha fusjonert til å forme kromosom 2 hos mennesket. Det var en størrelsesforskjell på ca. 10% eller 24 millioner baser i den foreslåtte fusjonen. Vi kjenner nå til at de tilsmussede bandene en refererte til biokjemisk, tilsvarer GC og AT %-andeler i DNA, ikke spesifikke gener eller gen-grupper. De mange genene i området som omgir den antatte fusjonen, utgjør 614.000 baser som ikke finnes i sjimpanse, og deler ikke komparativ likhet med kromsom 2A eller 2B hos sjimpanse.
Opprinnelig idé for fusjonsmodellen var basert på delt bindings-mønstre observert under lys-mikroskopi for kjemisk tilsmussede kromosomer. Prosessen er basert på isolering av celler som gjennomgår kondensering under mitose (celledeling), som tilsmusses med et stoff som binder til DNA, og produserer bindemønstre (karyotyping). Da dette ble gjort for sjimpanser, var det mange små kromosomer som delte mange band lik menneskets kromosom 2, og ble deretter postulert å ha fusjonert til å forme kromosom 2 hos mennesket. Det var en størrelsesforskjell på ca. 10% eller 24 millioner baser i den foreslåtte fusjonen. Vi kjenner nå til at de tilsmussede bandene en refererte til biokjemisk, tilsvarer GC og AT %-andeler i DNA, ikke spesifikke gener eller gen-grupper. De mange genene i området som omgir den antatte fusjonen, utgjør 614.000 baser som ikke finnes i sjimpanse, og deler ikke komparativ likhet med kromsom 2A eller 2B hos sjimpanse.  Det påståtte stedet for den evt. fusjonen er foreslått til kromosom 2, og DNA-segmentet ble først lokalisert og sekvensiert i 1991, omkring 9 år etter fusjonen først ble foreslått ved sammenlignende studier. Men den antatte fusjonen ble noe av en overraskelse for forskere, av det som syntes som en korrupt telomer fusjon. Ut fra hva de kjente av fusjonsdata fra levende pattedyr, hadde de forventet noe annet enn dette. Også tapet av 10% informasjon, var vanskelig å forklare.
Det påståtte stedet for den evt. fusjonen er foreslått til kromosom 2, og DNA-segmentet ble først lokalisert og sekvensiert i 1991, omkring 9 år etter fusjonen først ble foreslått ved sammenlignende studier. Men den antatte fusjonen ble noe av en overraskelse for forskere, av det som syntes som en korrupt telomer fusjon. Ut fra hva de kjente av fusjonsdata fra levende pattedyr, hadde de forventet noe annet enn dette. Også tapet av 10% informasjon, var vanskelig å forklare.  den hypotetiske kromosom 2-fusjonen de forventede karakteristika av en ende-til-ende fusjon for to kromosomer? For å si det kort: dataene passer ikke. Sekulære evolusjonsforskere har uttalt dette faktum i sine forsknings rapporter (se under). Kanskje en av de mest avslørende var en nøye DNA-sekvens analyse, av fusjonsområdet av molekyl-genetikere, der de merket seg at den fusjonerte DNA-sekvensen var ekstremt degenerert, ut fra sitt hypotetiske opphav. 6 millioner år er overgang fra primater til menneske antatt å vare. I det store tidsdypet, er det kun en kort periode. Sammenlignet med en opprinnelig fusjon av samme signatur-størrelse 8798 baser), så er det formodede fusjonsstedet bare ca. 88% likt. Noen har kommentert dette slik: "hode-til-hode arrayer av repetisjoner ved fusjonsstedet har degenert betydelig, fra de nær perfekte arrayene (TTAGGG) som finnes i telomerer" Og: "om fusjonen inntraff innen telomer-repeterende arrayer, for mindre enn 6 millioner år siden: hvorfor er arrayene på fusjonsstedet så degenererte?
den hypotetiske kromosom 2-fusjonen de forventede karakteristika av en ende-til-ende fusjon for to kromosomer? For å si det kort: dataene passer ikke. Sekulære evolusjonsforskere har uttalt dette faktum i sine forsknings rapporter (se under). Kanskje en av de mest avslørende var en nøye DNA-sekvens analyse, av fusjonsområdet av molekyl-genetikere, der de merket seg at den fusjonerte DNA-sekvensen var ekstremt degenerert, ut fra sitt hypotetiske opphav. 6 millioner år er overgang fra primater til menneske antatt å vare. I det store tidsdypet, er det kun en kort periode. Sammenlignet med en opprinnelig fusjon av samme signatur-størrelse 8798 baser), så er det formodede fusjonsstedet bare ca. 88% likt. Noen har kommentert dette slik: "hode-til-hode arrayer av repetisjoner ved fusjonsstedet har degenert betydelig, fra de nær perfekte arrayene (TTAGGG) som finnes i telomerer" Og: "om fusjonen inntraff innen telomer-repeterende arrayer, for mindre enn 6 millioner år siden: hvorfor er arrayene på fusjonsstedet så degenererte?  liten signatur, for telomer-sekvensen.
liten signatur, for telomer-sekvensen. Lokaliseringen til fusjonsområdet inni et funksjonelt og høyt uttrykt gen, assosiert med uttrykk i vidt omfattende cellekategorier, benekter sterkt idéen at det er et biprodukt av en fusjon som aldri inntreffer i naturen. Komplekse, funksjonelle gener kan ikke dannes av katastrofale kromosom-fusjoner.
Lokaliseringen til fusjonsområdet inni et funksjonelt og høyt uttrykt gen, assosiert med uttrykk i vidt omfattende cellekategorier, benekter sterkt idéen at det er et biprodukt av en fusjon som aldri inntreffer i naturen. Komplekse, funksjonelle gener kan ikke dannes av katastrofale kromosom-fusjoner. Enda et annet hovedproblem ved fusjonsmodellen er mangel på bevis for en kryptisk sentromer lokasjon. Sentromerer er den sentrale delen av et kromosom, som spiller en nøkkelrolle i kontroll ved celledeling og gen-stabilitet. Om det var en hode-til-hode fusjon av to kromosomer -som nevnt ovenfor, ville det være to centrosomer i det fusjonerte kromosomet. Nå er det dårlig nytt for et kromosom, den kan bare ha én. Én av centrosomene måtte umiddelbart elimineres, eller i det minste få stilnet funksjonen for celledeling, ellers ville kromosomet selv-destrueres. Interessant nok er tegnene på en kryptisk sentromer i kromosom 2 enda svakere enn for et telomer-rikt fusjonssted. Det kan også bemerkes at angivelig sted for en kryptisk sentromer, er på feilaktig sted, i forhold til fusjonsmodellen.
Enda et annet hovedproblem ved fusjonsmodellen er mangel på bevis for en kryptisk sentromer lokasjon. Sentromerer er den sentrale delen av et kromosom, som spiller en nøkkelrolle i kontroll ved celledeling og gen-stabilitet. Om det var en hode-til-hode fusjon av to kromosomer -som nevnt ovenfor, ville det være to centrosomer i det fusjonerte kromosomet. Nå er det dårlig nytt for et kromosom, den kan bare ha én. Én av centrosomene måtte umiddelbart elimineres, eller i det minste få stilnet funksjonen for celledeling, ellers ville kromosomet selv-destrueres. Interessant nok er tegnene på en kryptisk sentromer i kromosom 2 enda svakere enn for et telomer-rikt fusjonssted. Det kan også bemerkes at angivelig sted for en kryptisk sentromer, er på feilaktig sted, i forhold til fusjonsmodellen.  Konklusjon: dårlige data gir ingen fusjon
Konklusjon: dårlige data gir ingen fusjon